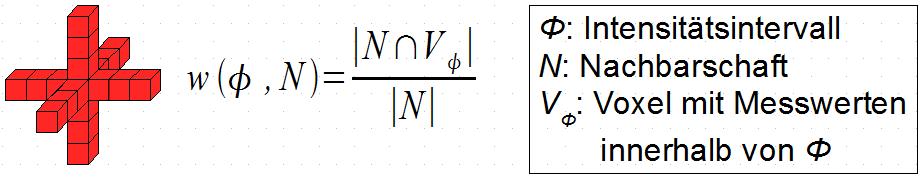Orts- und zeitabhängige Transferfunktionen für Direct Volume Renderingin der medizinischen Datenvisualisierung
Masterarbeit von Eugen Dyck
Motivation
Mittels CT- oder MRT-Aufnahmen ist es heute möglich nicht nur zweidimensionale Bilder von Patienten (wie etwa bei einer gewöhnlichen Röntgenaufnahme) zu erzeugen, sondern auch dreidimensionale. Die Computer-Tomographie beispielsweise erzeugt mehrere sukzessive Schichtbilder, die gemeinsam als Volumen dargestellt werden können. Mit aktueller Grafik-Hardware ist es möglich dieses Volumen auf einem handelsüblichen PC direkt darzustellen.
Mittels mehrerer an das EKG gekoppelter CT Aufnahmen (Frames) ist es auch möglich Formänderungen etwa des Herzens über die Zeit festzuhalten. Solche zeitabhängigen (vierdimensionalen) Datensätze erlauben die Untersuchung der Dynamik des Herzens oder auch die Analyse des Blutflusses in den Herzkammern. Sie können auch zur dynamischen Visualisierung eines schlagenden Herzens herangezogen werden.
Damit ein Arzt einen die Darstellung eines Datensatz mit mehr als zwei Dimensionen zur Diagnose verwenden kann, ist eine interaktive Exploration und Bestimmung der einzeln darzustellenden Gewebearten nötig. Soll in etwa das Skelett eines Patienten sichtbar gemacht werden, müssen die das Skelett umgebenden Gewebestrukturen, wie Muskel, Bindegewebe, Haut, etc., ausgeblendet werden. Das Herausstellen (Klassifizieren) von Gewebestrukturen leisten unter anderem so genannte Transferfunktionen.
Motivation dieser Arbeit ist die Frage nach der Relevanz der orts- und zeitabhängigen Lage eines Gewebes für seiner Klassifizierung.
Ziel der Arbeit
Am Lehrstuhl für Computergrafik, Visualisierung und Bildverarbeitung an der Universität Paderborn wurden bisher zweidimensionale Transferfunktionen zur Klassifizierung verwendet. Als Eingabedimensionen dienen zum einen der gemessene Datenwert aus der CT-Aunahme selbst und zum anderen der daraus berechnete absolute Gradient, der den Betrag der Datenwertänderung in der unmittelbaren Nachbarschaft angibt. Ein allgemeines Problem bei Transferfunktionen ist, dass verschiedene Gewebearten gleiche Eigenschaften aufweisen können. So wird mit Kontrastmittel angereichertes Blut im CT mit dem selben Wert wie Teile des Skeletts gemessen. Eine Separation dieser Gewebearten ist dann allein über den gemessenen Datenwert nicht möglich. Mit Hilfe weiterer Dimensionen wird nun versucht eine Unterscheidung solcher Gewebe vorzunehmen.
Ziel dieser Arbeit ist die Untersuchung der Verwendung von „Ort“ und „Zeit“ als Eingabeparameter der Transferfunktion und der daraus resultierenden Qualität der Klassifizierung. Die entworfenen Methoden für die Ortsabhängigkeit sollen direkt mit dem bisher verwendeten Gradienten als Eingabeparameter verglichen werden. Für zeitabhängige Datensätze soll geprüft werden wie sich diese allgemein klassifizieren lassen. Die konkrete Frage ist hierbei, ob jeder Frame einzeln klassifiziert werden muss, ob die Klassifizierung eines Frames global auf alle weiteren Frames angewendet werden kann oder ob Framegruppen klassifiziert werden müssen, um in jedem Frame die selbe Gewebestruktur zu selektieren.
Realisierung
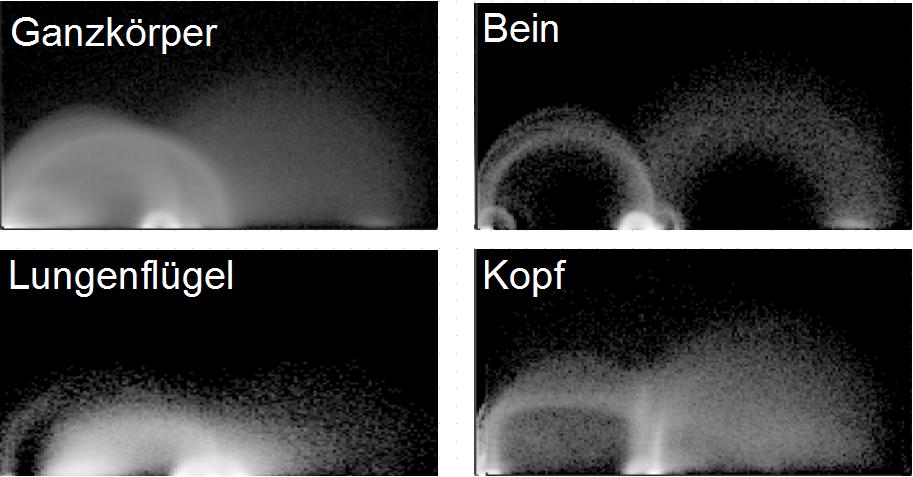
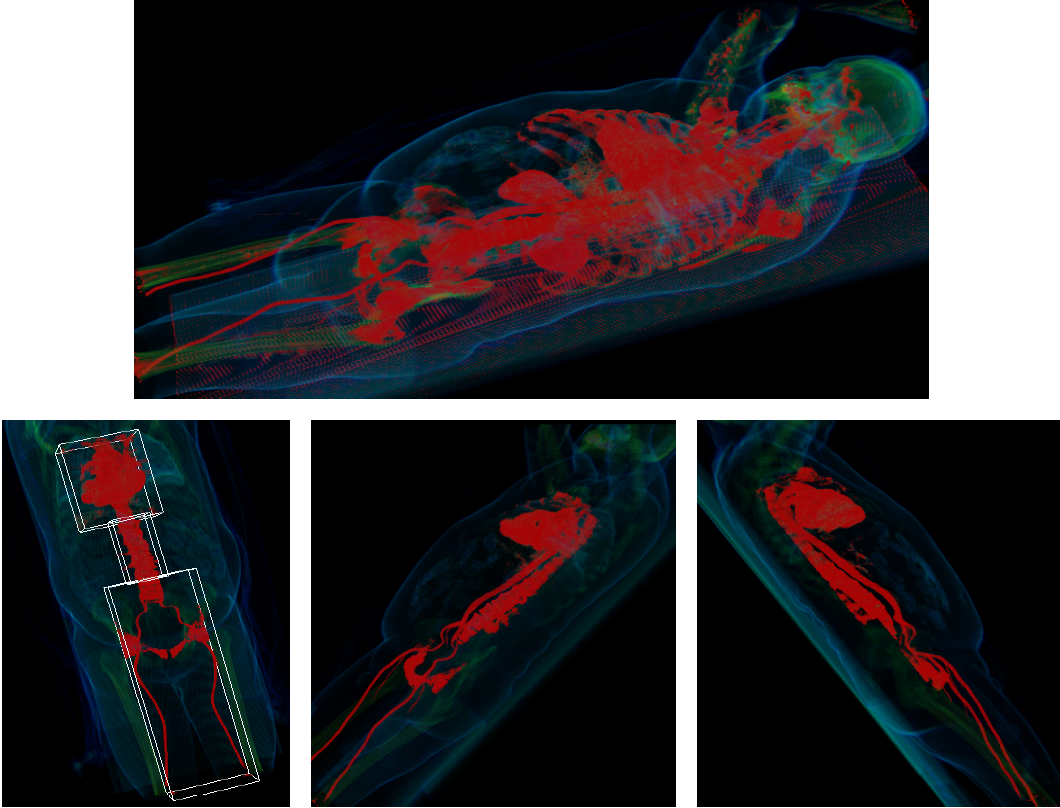
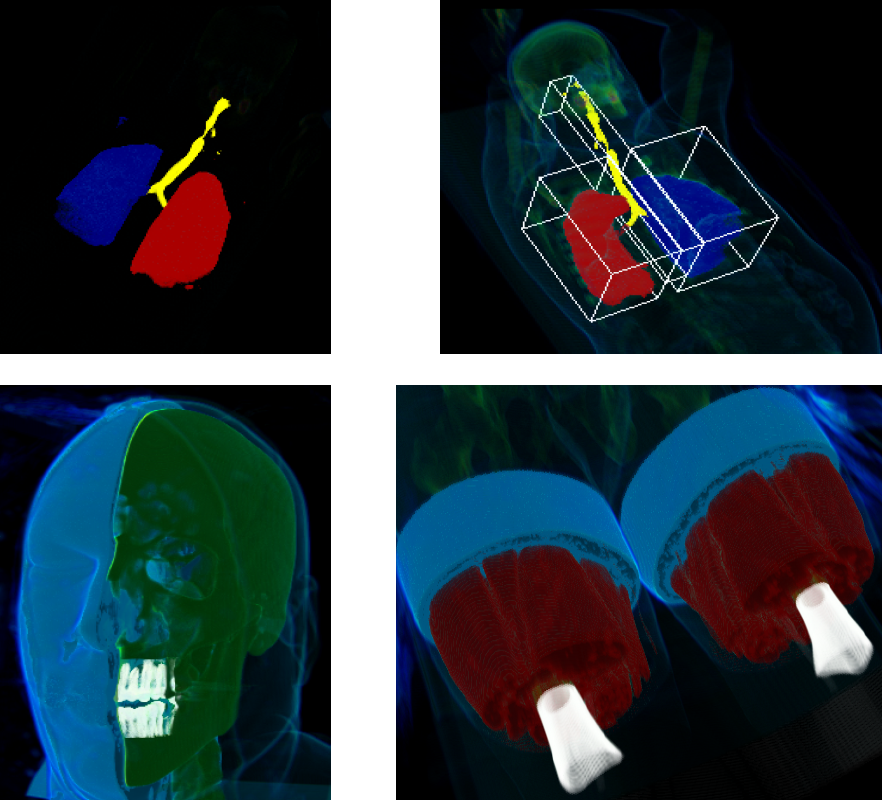
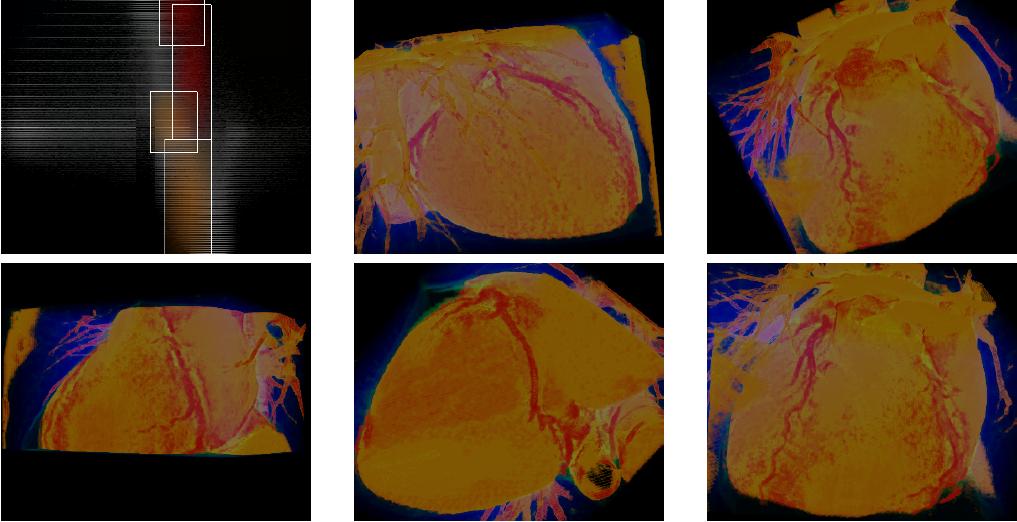
Zur Erstellung der Transferfunktion werden Histogramme verwendet, die die Werteverteilung pro Eingabedimension darstellen. Bei komplexen Datensätzen, etwa bei einem Ganzkörper-CT, überlagern sich die Werte verschiedener Gewebe, da ihr Schwächungskoeffizient in Bezug auf Röntgenstrahlung gleich ist. Dies erschwert die Analyse des Histogramms und damit die Erstellung der Transferfunktion. Um dies zu vereinfachen, führt diese Arbeit das Boundingbox-Konzept ein. Hierbei wird aus dem globalen Volumen ein Subvolumen zur Berechnung des Histogramms herangezogen, das das zu klassifizierende Gewebe einschließt. Jeder Voxel erhält somit eine Zugehörigkeit zu dem globalen oder einem örtlich begrenzten Volumen. Pro Datensatz können mehrere Subvolumen erzeugt und eine Separation gleicher Gewebeeigenschaften auf Grund ihrer örtlichen Lage vorgenommen werden. Hier kommt also die Ortsinformation zusätzlich zum Datenwert und dem Gradienten als Eingabeparameter dazu.
Zusätzlich zum Boundingbox-Konzept wird das Nachbarschaftsgewicht als Kriterium zur Klassifizierung eingeführt. Dies ersetzt den Gradienten als Eingabeparameter der Transferfunktion. Das Nachbarschaftsgewicht berechnet sich aus der Anzahl aller Voxel mit gleichem Datenwert wie dem des untersuchten innerhalb einer definierten Nachbarschaft. Die Nachbarschaft ist dabei ein dreidimensionales Kreuz, dessen Radius und weitere Parameter vom Benutzer eingestellt werden können. Damit wird die Selektion von besonders „kleinen Organen“ oder besonders „großen“ konfigurierbar. Die Ortsabhängigkeit also ist hierbei gekoppelt an den Datenwert des untersuchten Voxels. Sie ist eine Abhängigkeit von der definierten Ausdehnung der Nachbarschaft, betrachtet also nicht die absolute Lage im Raum, sondern die relative, und zwar für einen gegebenen Datenwert.
Für zeitabhängige Herz-Datensätze wurde eine Klassifizierungsmöglichkeit pro Frame geschaffen. Der Benutzer hat dabei die Möglichkeit die durchgeführte Klassifizierung auf frei wählbare Frames anzuweiten, insbesondere auf alle Frames gleichzeitig. Die verwendete Transferfunktion ist dabei ebenfalls frei wählbar. Es kann sich also um bisher verwendete Datenwert-Gradient- oder Datenwert-Nachbarschaftsgewicht-Transferfunktionen handeln. Als Hilfswerkzeug werden verschiedene Zeithistogramme zur besseren Übersicht verwendet.
Im Ergebnis zeigt sich, dass das Boundingbox-Konzept erhebliche Vorteile bei der Klassifizierung mit sich bringt. So ermöglicht es die Separation gleicher Gewebe auf Grund ihrer örtlichen Lage und die vereinfachte Analyse der Histogramme. Das Konzept der Nachbarschaftsgewichte erzeugt im Vergleich zu dem gradientenbasierten Ansatz deutlichere Separation, die Nachbarschaftsgewichte müssen jedoch pro zu klassifizierendes Gewebe neu berechnet werden. Dies geschieht zwar mit vertretbarem Konfigurationsaufwand aber eben mit Aufwand, das Gewöhnung braucht. Besonders herauszustellen ist hier die Separation von Koronargefäßen von der dickeren Aorta und den Blutansammlungen in den Ventrikeln, was mit dem gradientenbasierten Verfahren nicht gelingt.
Für die zeitabhängigen Herz-Datensätze hat sich gezeigt, dass die Klassifizierung eines Frames mittels Datenwert-Gradient-Transferfunktion auf alle weiteren Frames ohne feststellbare Verluste ausgeweitet werden kann.