Realisierung einer texturbasierten Transferfunktion zur Visualisierung medizinischer CT/PET-Volumendaten
Bachelorarbeit von Annette C. Keusch und Dominik Lüke
Motivation
Medizinisches Fachpersonal sieht sich in zunehmendem Maße mit immer größeren Mengen medizinischer Bilddaten konfrontiert, die es zu diagnostischen oder therapeutischen Zwecken auszuwerten gilt. Die damit verbundene Inspektion der Bilddaten bindet wertvolle Zeitressourcen, die an anderer Stelle fehlen. Eine rechnergestützte, automatische Visualisierung der Bilddaten, die den diagnostischen Prozess durch eine geeignete Darstellung der Bildinhalte unterstützt, kann somit zu einer wesentlichen Entlastung beitragen.
Insbesondere der differenzierten Visualisierung unterschiedlicher Gewebetypen kommt eine tragende Rolle zu. Hierbei stoßen gängige Verfahren wie z. B. ein- oder zweidimensionale Transferfunktionen, die Gewebe anhand der bloßen Intensitätswerte der Voxel des Volumenbildes bzw. deren Gradienten zu unterscheiden suchen, schnell an ihre Grenzen: im Falle von unterschiedlichen Geweben, die im Bildvolumen durch gleiche oder sich überlappende Intensitätswertebereiche repräsentiert werden, ist eine diskriminative Darstellung der Gewebe auf diesem Wege nicht möglich.
Zielsetzung
In dieser Bachelorarbeit soll untersucht werden, ob der Nachteil der bisher eingesetzten Transferfunktionen, Gewebe mit gleichem Grauwert und Gradienten nicht differenziert visualisieren zu können, durch das Konzept der texturbasierten Transferfunktionen [1] ausgeglichen werden kann. Unter Textur wird in diesem Kontext die räumliche Verteilung bzw. Anordnung von Intensitätswerten im Volumendatensatz verstanden.
Hierzu soll das Konzept der texturbasierten Transferfuntkionen in die Software Volume Studio implementiert werden. Darauf aufbauend soll ein weiteres Verfahren implementiert werden, mit dessen Hilfe es möglich sein soll, zwei Strukturen innerhalb eines Volumendatensatzes getrennt zu visualisieren. Die Effektivität dieses Verfahrens soll anschließend durch eine qualitative und quantitative Evaluation untersucht werden.
Realisierung
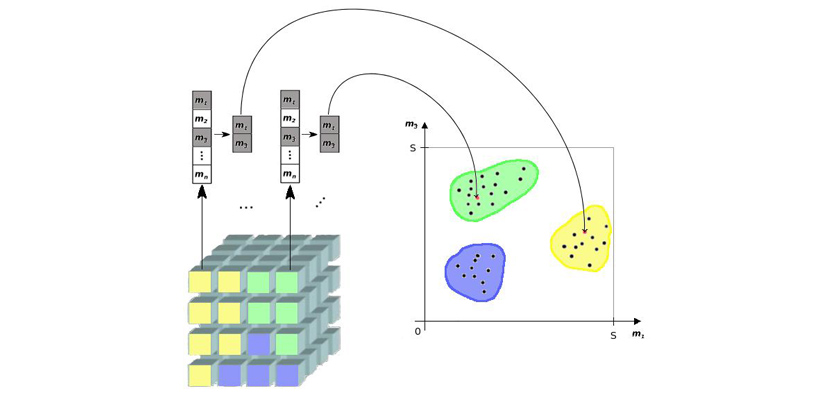
Das in [1] vorgestellte Konzept der texturbasierten Transferfunktionen wurde in Form eines automatischen Klassifikationsverfahrens umgesetzt und in der Software Volumestudio als Plugin implementiert. Im Zuge des Verfahrens wird dabei zunächst eine geeignete Partitionierung des Volumendatensatzes in Subvolumina vorgenommen, für die jeweils eine quantitative Texturanalyse erfolgt. Als deren Ergebnis steht die Beschreibung der lokalen Textur eines jeden Subvolumens durch einen Vektor von skalaren Merkmalen. Die anschließende Klassifikation des Volumendatensatzes erfolgt mit Hilfe des Clusteralgorithmus k-Means auf den ermittelten Merkmalsvektoren der Subvolumina im Merkmalsraum (bzw. auf einer beliebig wählbaren Untermenge von Merkmalen). Sie basiert somit alleinig auf lokalen texturellen Eigenschaften.
Durch Zuweisung individueller Farben und Opazitäten zu den gefundenen Gruppen kann eine texturbasierte Visualisierung der Volumendaten realisiert werden. Dabei werden die Voxel eines Subvolumens mit der Gruppe assoziiert, zu welcher der Texturmerkmalsvektor des Subvolumens zugeordnet wurde (siehe Abb. 1).
Für den Schritt der Texturanalyse wurden Metriken der Statistik erster und zweiter Ordnung als Texturmerkmale im Verfahren integriert. Ihre Auswahl orientierte sich an [1] sowie an den Ergebnissen einer einschlägigen Literaturrecherche.
Aufbauend auf diesem generellen Verfahren wurde gemäß der Zielsetzung ein zweites Verfahren implementiert, welches durch eine automatisch bestimmte Gewichtung der Metriken die Unterschiede zweier im Bildvolumen selektierter Gewebestrukturen im Klassifikationsprozess maximiert, um potentiell eine getrennte Visualisierung der Gewebe zu ermöglichen.
Um die Ergebnisse der rechenintensiven Texturanalyse bei erneuten Klassifikationen auf unveränderter Partitionierung ein und desselben Volumendatensatzes wiederholt nutzen zu können, wurde eine entsprechende Möglichkeit zur Persistierung der ermittelten Texturmerkmale realisiert. Zur Performancesteigerung während des Klassifikationsprozesses wurde zudem eine besondere Version von k-Means implementiert, welche die Anzahl kostenträchtiger Distanzberechnungen unter Ausnutzung von Erkenntnissen auf Grundlage der Dreiecksungleichung erheblich reduziert [2].
Evaluation
Zur Feststellung der Effektivität des zweiten implementierten Verfahrens wurden eine quantitative und eine qualitative Evaluation durchgeführt. Bei beiden Evaluationen bestand die Zielsetzung darin, Herzgewebe von Knochengewebe möglichst genau zu trennen.
Die quantitative Evaluation hat anhand einer statistischen Auswertung der klassifizierten Voxel gezeigt, dass Herzgewebe nicht eindeutig von Knochengewebe getrennt werden kann.
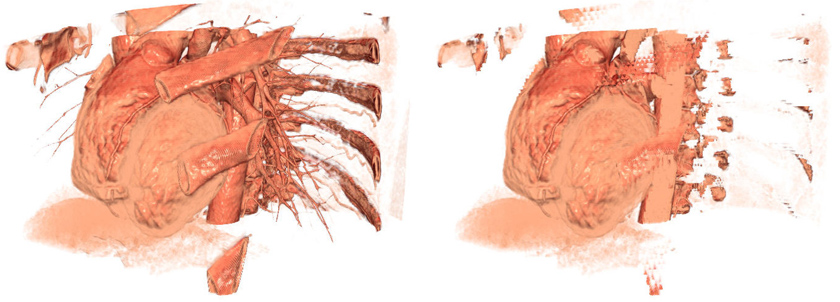
Bei der qualitativen Evaluation wurden konventionelle 2D-Transferfunktionen mit dem zweiten implementierten Verfahren kombiniert. Als Grundlage diente der durch konventionelle 2D-Transferfunktionen hervorgehobene Herzmuskel samt Knochengewebe (siehe Abb. 2 - links). Ausgehend von dieser Darstellung wurde das durch das zweite implementierte Verfahren erkannte Knochengewebe entfernt. Hierbei wurde ein Großteil des Knochengewebes und ein minimaler Teil des Herzmuskels entfernt (siehe Abb. 2 - rechts).
Entgegen dem mäßigen Ergebnis der quantitativen Evaluation wurde bei der qualitativen Evaluation ein gutes Ergebnis erzielt.
Literatur
[1] CABAN, Jesus J. ; RHEINGANS, Penny: Texture-based Transfer Functions for Direct Volume Rendering. In: IEEE Transactions on Visualization and Computer Graphics 14 (2008), Nr. 6, S. 1364–1371. – ISSN 1077–2626
[2] ELKAN, Charles: Using the Triangle Inequality to Accelerate k-Means. In: ICML, 2003, S. 147–153