Verwendung des Scale Space zur Darstellung medizinischer Volumendaten
Bachelorarbeit von Nico Bredenbals
Motivation
In der heutigen Zeit ermöglicht leistungsstarke Hardware die Echtzeitvisualisierung von dreidimensionalenVolumendaten. Eine der größten Herausforderungen, mit denen wir uns im Rahmen der Volumenvisualisierung konfrontiert sehen, ist daher nicht mehr das bloße Darstellen gewonnener Daten, sondern das Herausstellen der Regionen und Merkmale von Interesse. Während für einen Kardiologen hauptsächlich Herz und Herzgefäße von Bedeutung sind, benötigt ein Kieferchirurg den Kieferknochen, Zähne, Nervenbahnen und Blutgefäße zum Planen seiner Arbeit. Ein Sicherheitsbeamter wiederum möchte alle vorhandenen Merkmale, möglichst deutlich von einander abgegrenzt, visualisieren. Geräte zur Bilderfassung, wie zum Beispiel Computer-, Magnetresonanz- oder Positronenemissionstomographen, messen die Intensität einer physikalischen Eigenschaft eines Volumens, im Fall eines CT zum Beispiel die Dichtewerte des zu betrachtenden Objekts.Das führt zu Problemen: Häufig haben Strukturen, die vom Anwender unterschieden werden wollen, sehr ähnliche Intensitätswerte. So liegen zum Beispiel Milz und Niere bezüglich ihrer Dichte im Bereich der Leber, können allein anhand der Dichte also nicht unterschieden werden. Ein ähnliches Problem besteht bei der sehr gut durchbluteten Knochenhaut, die nicht von Arterien unterschiedenwerden kann. Nimmt man allerdings andere Metriken als den Dichtewert hinzu, wie zum Beispiel den Grad der Krümmung, den Gradienten oder die Größe eines Merkmals, lassen sich genannte Beispiele sehr wohl unterscheiden. Die Zielsetzung moderner Volumenvisualisierung ist also, dem Anwender Metriken an die Hand zu geben, die es ihm ermöglichen, die Gebiete, die für ihn von Interesse sind, schnell und intuitiv hervorheben zu können.
Zielsetzung
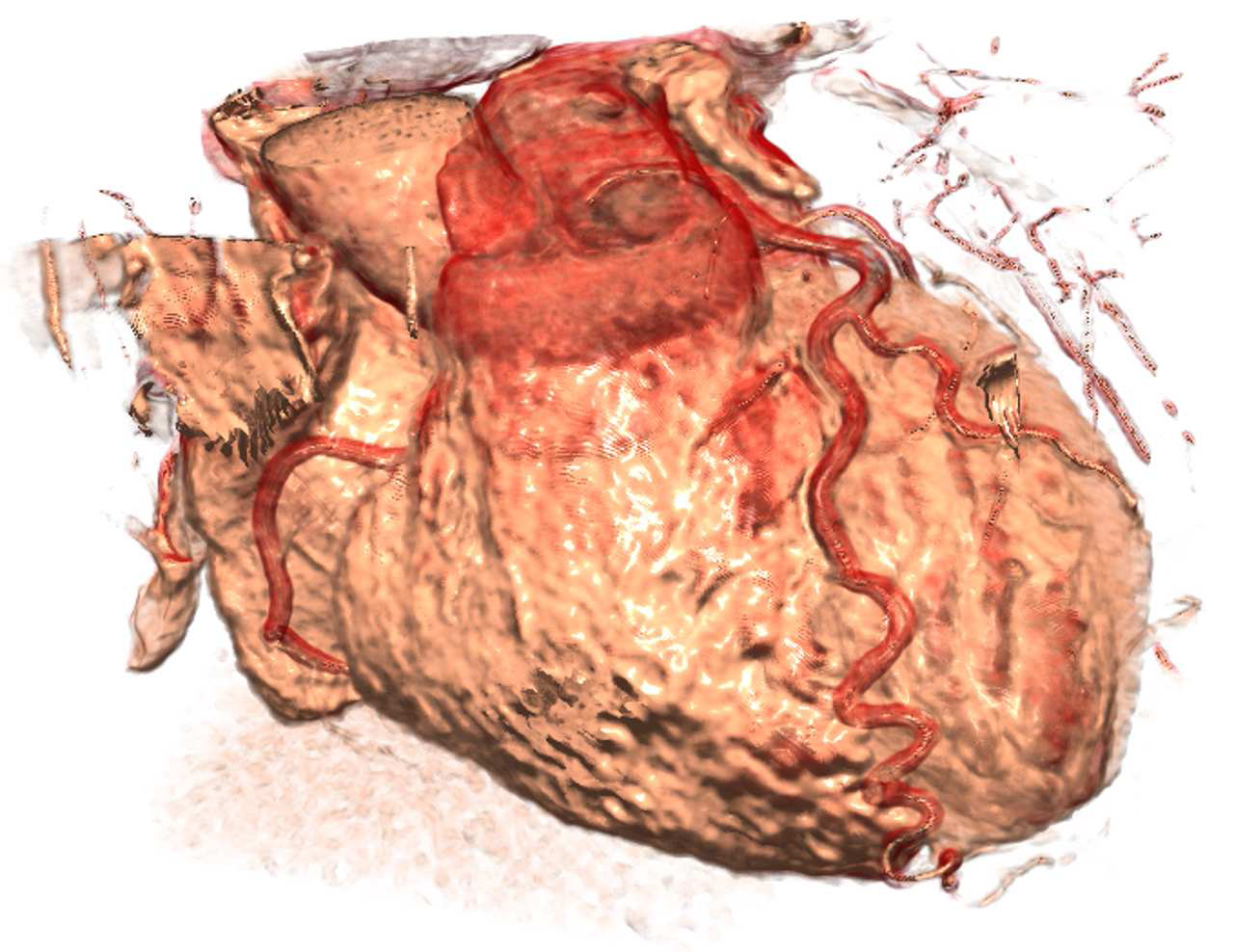
In dieser Arbeit wird die Größe als Metrik zur Klassifikation vorgestellt. Das hierfür verwendete Verfahren ist in der Lage Mittelpunkte von Merkmalen zu erkennen, und den Merkmalen eine relative Größe zu zuordnen. Hierzu werden im Rahmen der Arbeit zunächst die Grundlagen, sowohl medizinisch als auch im Bezug auf das Volumenrendering, erläutert, um darauf aufbauend die mathematische Fundierung zu erklären und das Verfahren als Erweiterung für die Software Volume Studio zu implementieren. Die Ergebnisse der Implementierung werden im Anschluss anhand verschiedener synthetischer und eines authentischen Datensatzes vorgestellt.
Realisierung
Es wird das von Carlos D. Correa und Kwan-Liu Ma in "Size Based Transfer Functions" vorgestellte Verfahren verwendet um Mittelpunkte zu erkennen, diesen eine relative Größe zuzuordnen, und basierend auf diesen Daten eine Abbildung zu erstellen die jedem Voxel eine relative Größe zuordnet. Zur Erkennung der Mittelpunkte und ihrer relativen Größe wird der aus der Bildverarbeitung bekannte "Scale Space" verwendet. Das Volumen wird hierbei mit Hilfe eines anisotropen Diffusionsverfahrens ausgewaschen. Die Anzahl der Iterationen die benötigt wird bis das Volumen ausgewaschen ist dient zur Ermittlung der Größe. Die Abbildung jedes Voxels auf die Größe des Merkmales zu dem es gehört wird mit Hilfe einer "multivariaten Interpolation" gelöst. Hierzu wurde zunächst eine naive iterative Variante implementiert. Diese wurde zu einer Laufzeiteffizienten, genäherten Variante erweitert.